Chapitre 7 Déploiement de modèles
L’ultime étape pour donner vie à un modèle est de le mettre en production.
Les exemples de déploiement de modèles sont omniprésents. Il peut s’agir de cas plus évidents comme l’obtention d’un score de crédit afin de déterminer l’éligibilité pour un prêt ou encore une soumission pour un produit d’assurance basé sur les caractéristiques de l’appliquant et du bien assuré. De manière plus large, ça comprend également des recommendations d’achat faites par un marchand en ligne, les utilitaires un appareil photo permettant la reconnaissance des visages ou un système de conduite autonome.
De par la diversité des cas de consommation des modèles, il n’existe pas de solution unique applicable à toutes les situations. Parmi les considérations, notons:
- nombre de requêtes qui devra être supporté: l’application est-elle destinée à un groupe d’employé ou à une masse de consommateurs localisés partout sur le globe?)
- besoins en latence: cherche-t-on simplement à assurer une expérience client dynamique ou doit-on exécuter un ordre d’achat dans un contexte d’arbitrage boursier à haute fréquence?
- les contraintes de la plateforme qui supoprtera les prédictions: par exemple un système de contrôle intelligent qui a des resources processeurs et mémoires limités
- besoins de stabilité dy système: est-ce qu’une défaillance ou une latence non-déterministe due aux collecteur de déchet pourrait entraîner une collision aérienne?
En présence de contraintes importantes sur les précédents facteurs, il peut être préférable d’avoir recours à des langages compilés (C/C++) ou encore plus près du matériel (CUDA) qu’un langage dynamique tel que R. Il n’en reste pas moins que pour un large éventail de contextes, une capacité de développement rapide combinée à des options de déploiement simples font de R une option intéressante pour le déploiement de modèle.
À très haut niveau, ce qu’on chercher à accomplir est de permettre à un usager extérieur à notre localisation, sans accès à R, d’accéder aux prédictions de notre modèle:
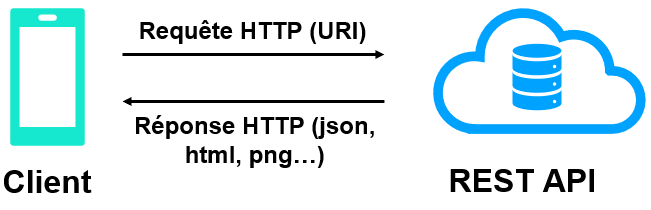
Nous verrons ici deux outils permettant de servir les résultats d’un modèle:
Chacun de ces outils permet de servir un modèle via un REST API, ce qui signifie que les échanges sont réalisés au travers du protocole http. Comme il s’agit d’un protocole omniprésent, il existe de nombreux outils disponibles pour en faciliter l’intégration dans différentes applications. Ça signifie que ces applications peuvent exploiter les capacités de modélisation de R sans qu’elles aient à intégrer R d’aucune façon. L’utilisation d’un modèle via un REST API devient donc agnostic du ou des langages utilisés à l’intérieur de cet API.
7.1 OpenCPU
L’unité de travail de OpenCPU repose sur la librairie. Pour pouvoir servir un modèle, on doit donc le structurer de manière à ce qu’il puisse être encapsulé à l’intérieur d’une librairie. Si l’approche peut paraître contraignante, elle incite à l’adoption d’une discipline dans la structure du code et de ses dépendances qui peut être salutaire à sa maintenance.
7.1.1 Débuter avec OpenCPU
Le déploiement d’un modèle se fait normalement à partir d’un serveur. Pour se familiariser avec l’outil ainsi que faciliter le développement, il est possible de rouler le serveur OpenCPU localement.
- Installer la librairie:
install.packages("opencpu")
- La charger:
library(opencpu) - Démarrer le serveur:
opencpu::ocpu_start_server()
Une fois le serveur démarré, il est désormais possible d’interagir avec celui-ci via le port local: http://localhost:5656/ocpu/test/ (localhost est ici équivalent à 127.0.0.1).
Une requête http peut alors être faite à ce serveur. La programme curl est communément utilisé pour ces requêtes et peut être utilisé à partir du Terminal dans RStudio (ou à partir de tout autre terminal) ou même à l’intérieur de R grâce à des librairies comme curl our httr:
$ curl http://127.0.0.1:5656/ocpu/library/stats/R/rnorm/json -d n=2
[-0.4539, 0.1959]Que s’est-il passé? Le serveur a reçu une requête pour la fonction rnorm situé dans stats/R/ avec n=2 comme paramètre. La segment /json a servi à demander à ce que le résultat soit retourné dans le format json. De fait, l’appel à la fonction a retourné un json contenant deux observations simulées d’une Normale(0,1).
Il a été possible de faire la précédente requête puisque par défaut, la librairie stats est accessible par OpenCPU, à l’instar de l’ensemble des librairies accessibles au niveau système. Pour servir un modèle prédictif, une librairie permettant de retourner les prédictions de ce modèle devra donc être développée et installée.
7.1.2 Bâtir un squelette de librairie
Avant d’illustrer le déploiement d’un modèle, nous allons présenter comment utiliser OpenCPU à partir d’un exemple le plus minimal possible.
La première étape est d’avoir le code structuré en une librairie. Une librairie peut être initialisée à l’aide de la commande:
usethis::create_package("~/<nom_de_la_librairie>/")
usethis::create_package("~/model.ocpu/")Alternativement, Rstudio offre la fonctionnalité à partir du menu: File -> New Project -> New Directory -> R Package.
Une librarie en R n’est qu’un projet qui contient un fichier DESCRIPTION, un fichier NAMESPACE ainsi qu’un dossier R/ à l’intérieur duquel se trouvent les codes R. Les fonctions d’initialisation ci-haut ne sont que des aides facilitant la création de cette structure.
Une fois la création du squelette complétée, une fonction de test peut être créée. Par exemple, avec le code suivant dans le fichier ./R/salut.R:
#' Salut
#' Une fonction qui salue.
#' @export
salut <- function() {
print("Salut tout le monde!")
}La librairie model.ocpu peut maintenant être bâtie. La documentation sera d’abord générée:
devtools::document()L’installation peut ensuite s’exécuter à l’aide du raccourci: ctrl-shift-B.
En démarrant une nouvelle session du serveur OpenCPU, il sera désormais possible d’intéragir avec la librairie model.ocpu nouvellement installée.
curl http://localhost:5656/ocpu/library/model.ocpu/R/salut/json -d ""7.1.3 Intégrer un modèle prédictif dans une librairie
Une librairie permettant de retourner des prédictions devra supporter les fonctionnalité suivantes: - Lire les informations relativement aux observations pour lesquelles une prédiction doit être retournée - Appliquer les possibles transformations utilisées dans la préparation des données sur lesquelle le modèle a été construit. - Effectuer la prédiction sur ces donnée à partir du modèle sélectionné pour la déploiement.
Comment rendre accessible le modèle entraîné à l’intérieur de la librairie? Un modèle peut être représenté comme la combinaison entre des paramètres et un algorithme décrivant comment de transformer les informations d’une observations en une prédiction. Lorsqu’un modèle est entraîné, l’objet résultant contient ces informations, de sorte que la fonction predict appliquée sur ce modèle permet d’obtenir les prédictions désirée.
L’approche la plus naturelle sera donc de sauvegarder le modèle désiré à l’intérieur de la librairie et de le rendre accessible aux fonctions de la librairie d’inférence.
La méthode recommandée consiste en la création d’un script générant un fichier .Rda qui contient les modèles et autres objets R nécessaires à l’inférence. Ce script sera localisé dans le dossier data-raw.
Par exemple, ce script sera le suivant pour rendre disponible le modèle développé à la section précédente:
source("../../../src/collecte/load-merging-data.R")
source("../../../src/init.R")
init_objects <- init(path_data = "../../../data/", path_objects = "../../../data/models/")
usethis::use_data(init_objects, internal = T, overwrite = T)Une fonction d’inférence peut maintenant être construite.
#' @export
bixikwargs <- function(start_date, start_station_code, is_member) {
# arranger en un data.table
dt_pred <- data.table(start_date, start_station_code, is_member)
dt_pred <- merge_data(dt_pred, init_objects$merging_data$data_stations)
data_pred <- preprocessing_main(copy(dt_pred), train_mode = FALSE, list_objects = init_objects)
data_pred_regression <- data_pred$data_regression
data_pred_classif <- data_pred$data_classif
duree = predict(init_objects$model_glm, as.matrix(data_pred_regression), s = "lambda.min")
meme_station = predict(init_objects$model_xgb, as.matrix(data_pred_classif)) > 0.5
return(list(duree = duree, meme_station = meme_station))
}curl http://localhost:5656/ocpu/library/model.ocpu/R/bixikwargs/json -d "start_date='2017-04-15 00:48'&start_station_code=6079&is_member=1"- Ajouter des fonctions et dépendances
7.2 Plumber
L’approche prise par Plumber repose sur l’ajout d’annotations au code. La technologie sous-jacente est similaire à OpenCPU, l’idée étant de convertir du code R en des services accessibles via le protocole HTTP.
7.2.1 Débuter avec Plumber
- Installer la librairie:
install.packages("plumber")
- La charger:
library(plumber)
Une exemple minimaliste est founi dans le dossier src/deploiement/model.plumber/plumber_ini.R. Ce code peut être servi comme un service plumber de la manière suivante:
pr <- plumber::plumb("src/deploiement/plumber/assets/plumber_ini.R")
pr$run(port=8985)Il est alors possible d’accéder au service:
curl http://127.0.0.1:8985/message?msg=Salut!7.2.2 Inférence de modèle prédictif
pr <- plumber::plumb("src/deploiement/plumber/assets/pred_bixi.R")
pr$run(port = 8985)7.3 Détails sur les requêtes HTTP
curl -d msg=Salut! -G http://127.0.0.1:8985/messagePasser les paramètres individuellement:
curl -X GET --data '{
"start_date": "2017-04-15 00:48",
"start_station_code": 6079,
"is_member": 1
}' http://localhost:8985/bixikwargscurl -X GET --data @src/deploiement/data_test_elements.json http://localhost:8985/bixikwargsPasser un fichier json en argument.
curl -X GET --data '{"data":[
{
"start_date": "2017-04-15 00:48",
"start_station_code": 6079,
"is_member": 1
}
]}' "http://localhost:8985/bixidata"curl -X GET --data @src/deploiement/data_test.json "http://localhost:8985/bixidata"La commande -X est utilisée pour forcer le remplacement des instructions par défaut. La méthode --data ou -d est en effet associés par défaut à une commande POST. Puisqu’on ne cherche ici qu’à retourner une réponse à une instruction, le verbe GET est approprié.
7.4 Déploiement avec Docker
Docker est une technologie permettant la containerization d’applications, faciliant leur portabilité sur différentes plateformes.
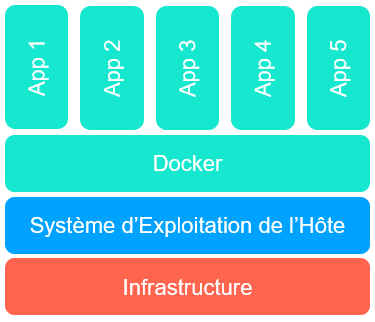
Une image Docker est formée de différentes couches contenant les libraries et configurations requises pour l’exécution de l’application. On peut construire une image à partir d’une racine très générique comme d’une installation générique de Ubuntu, ou encore d’une image intégrant déjà plusieurs fonctionnalités adaptées à notre domaine.
La recette pour la construction de ces images est spécifiée dans un fichier texte nommé Dockerfile.
docker build ./src/deploiement/plumber/ -t jeremiedb/dot-layer:plumberLancer une image.
docker run --rm -p 8080:8080 jeremiedb/dot-layer:plumber7.5 Intégration continue et webhook
Charest, Anne-Sophie. 2018a. “Analyse En Composantes Principales.” In STT-7330: Méthode d’analyse de Données. Québec, Canada: Université Laval.
———. 2018b. “Données Manquantes.” In STT-7330: Méthode d’analyse de Données. Québec, Canada: Université Laval.
Friedman, Jerome, Trevor Hastie, and Robert Tibshirani. 2001. The Elements of Statistical Learning. Vol. 1. 10. Springer series in statistics New York.
Hotelling, Harold. 1933. “Analysis of a Complex of Statistical Variables into Principal Components.” Journal of Educational Psychology vol 24: 417–41, 498–520.
James, Gareth, Daniela Witten, Trevor Hastie, and Robert Tibshirani. 2014. An Introduction to Statistical Learning: With Applications in R. Springer Publishing Company, Incorporated.
Khrunin, Denis V. AND Filippova, Andrey V. AND Khokhrin. 2013. “A Genome-Wide Analysis of Populations from European Russia Reveals a New Pole of Genetic Diversity in Northern Europe.” PLOS ONE 8 (3). Public Library of Science: 1–9. doi:10.1371/journal.pone.0058552.